3. “party” Package
3.1“party”패키지의 옵션
“party” 패키지의 ctree 함수가 Classification Tree를 학습할 때 사용자가 지정할 수 있는 옵션의 종류와 의미는 다음과 같습니다.
ctree 함수: ctree(formula, data, subset = NULL, weights = NULL, controls = ctree_control(), xtrafo = ptrafo, ytrafo = ptrafo, scores = NULL)
| 옵션 | 설명 |
|---|---|
| subset | 데이터에서 특정 행만을 선택해 사용하도록 한다. |
| controls | ctree_control을 통해 tree 구축의 세부적 사항을 설정한다. minsplit: split하기 전 필요한 weight 합의 최소값 minbucket: leaf node의 weight 합의 최소값 mtry: random forest와 비슷한 알고리즘에서 사용할 input variable의 개수 maxdepth: 최종 tree의 최대 depth (root node는 0으로 카운트된다.) |
| xtrafo | input변수에 적용되는 함수 설정 |
| ytrafo | y변수에 적용되는 함수 설정 |
3.2 옵션의 변화에 따른 Classification Tree들의 차이점 및 Best Model 선정
3.2.1 minsplit = 10, 50, 100
위의 rpart 패키지에서 minsplit과 maxdepth 모두 tree의 depth를 조정하여 비슷한 결과가 나옴을 확인했기 때문에 여기서는 minsplit만을 조정했습니다.
#3. Party package____________________________________________________________________________________
library(party)
Perf.Table <- matrix(0, nrow = 1, ncol = 6)
colnames(Perf.Table) <- c("TPR", "Precision", "TNR", "Accuracy", "BCR", "F1-Measure")
#3.2.1 minsplit = 10, 50, 100
for (i in c(10, 50, 100)){
heart.party <- ctree(heart.target ~., data = heart.trn,control = ctree_control(minsplit = i) )
plot(heart.party)
# Prediction
heart.prey <- predict(heart.party, heart.tst, type = "response")
heart.cfm <- table(heart.tst$heart.target, heart.prey)
heart.cfm
Perf.Table<- rbind(Perf.Table, perf_eval(heart.cfm))
Perf.Table
}
Perf.Table
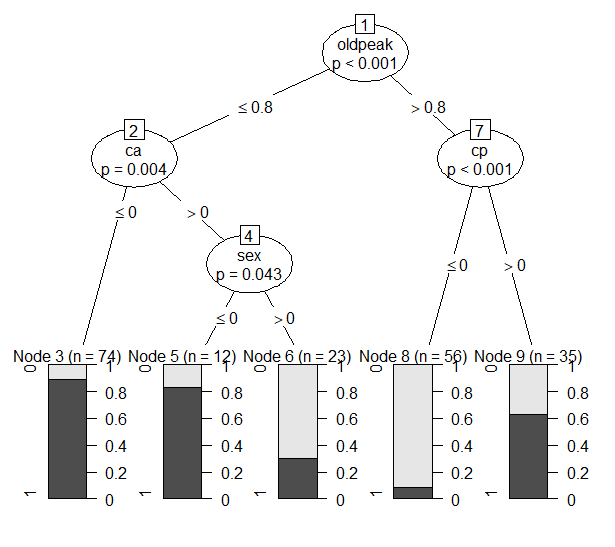
Figure 9 minsplit = 10
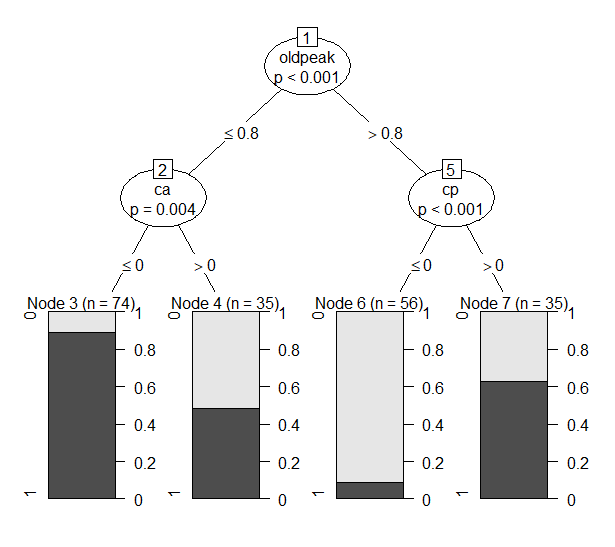
Figure 10 minsplit = 50
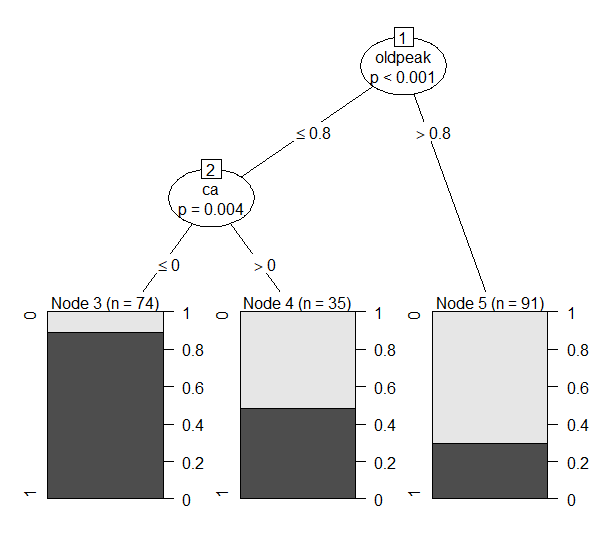
Figure 11 minsplit = 100
| 구분 | TPR | Precision | TNR | Accuracy | BCR | F1-Measure |
|---|---|---|---|---|---|---|
| minsplit = 10 | 0.763636 | 0.763636 | 0.729167 | 0.747573 | 0.746203 | 0.763636 |
| minsplit = 50 | 0.690909 | 0.745098 | 0.729167 | 0.708738 | 0.70978 | 0.716981 |
| minsplit = 100 | 0.454545 | 0.757576 | 0.833333 | 0.631068 | 0.615457 | 0.568182 |
- Minsplit이 커짐에 따라 단순한 tree가 구축되었다.
- Minsplit = 10으로 설정했을 때는 4번의 split이 있었고 5개의 terminal node가 생성되었다. 가장 먼저 사용된 변수는 oldpeak로 이후 ca, cp, sex 변수가 사용되었다. Node 6,8은 impurity가 낮은 편이었지만 node 3,5,9는 impurity가 0.5에 가까워 보였다. 성능 측면에서는 minsplit이 50일때와 100일때보다 훨씬 좋은 성능을 보였다. TNR을 제외한 모든 성능 지표에서 1위를 차지했으며 특히 Accuracy와 BCR, TPR에서 다른 모델과 차이가 많이 났다.
- Minsplit = 50으로 설정했을 때는 3번의 split을 통해 4개의 leaf node가 생성되었다. 역시 가장 먼저 사용된 변수는 oldpeak였고, 이후 ca와 cp변수가 사용되었다. Minsplit = 10일 때와 비교해 왼쪽 노드가 pruning 된 형태임을 알 수 있다. 성능 지표들은 대부분 2위를 차지했다. Node 3, 6은 impurity가 낮지만 node 4, 7은 impurity가 높은 편이었다.
- Minsplit = 100으로 설정했을 때는 2번의 split이 있었고 3개의 leaf node가 생성되었다. Minsplit = 50인 tree에서 오른쪽 노드가 pruning된 형태임을 알 수 있다. 하지만 너무 단순하기 때문에 분류를 제대로 못하는 것으로 나타났다. Accuracysms 0.63, TPR은 0.4545로 다른 모델들과 비교해 현저히 낮은 수치를 나타냈다. F1 역시 0.5681로 매우 낮았다. TNR만 0.8333으로 가장 높은 값을 보였는데, 나머지 지표들이 너무 낮아 좋지 않은 모델이라고 판단했다.
3.2.2 mtry = 3,4,5
Mtry는 선택되는 변수의 개수를 나타낸다. 이를 변화시키며 그린 tree들은 다음과 같다.
#3.2.2 mtry = 3, 4, 5
Perf.Table <- matrix(0, nrow = 1, ncol = 6)
colnames(Perf.Table) <- c("TPR", "Precision", "TNR", "Accuracy", "BCR", "F1-Measure")
for (i in c(3, 4, 5)){
heart.party <- ctree(heart.target ~., data = heart.trn,
control = ctree_control(mtry = i) )
plot(heart.party)
# Prediction
heart.prey <- predict(heart.party, heart.tst, type = "response")
heart.cfm <- table(heart.tst$heart.target, heart.prey)
heart.cfm
Perf.Table<- rbind(Perf.Table, perf_eval(heart.cfm))
Perf.Table
}
Perf.Table
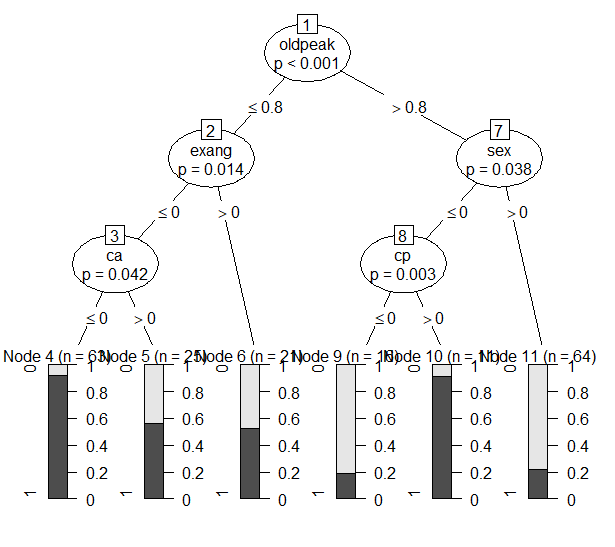
Figure 12 mtry = 3

Figure 13 mtry = 4
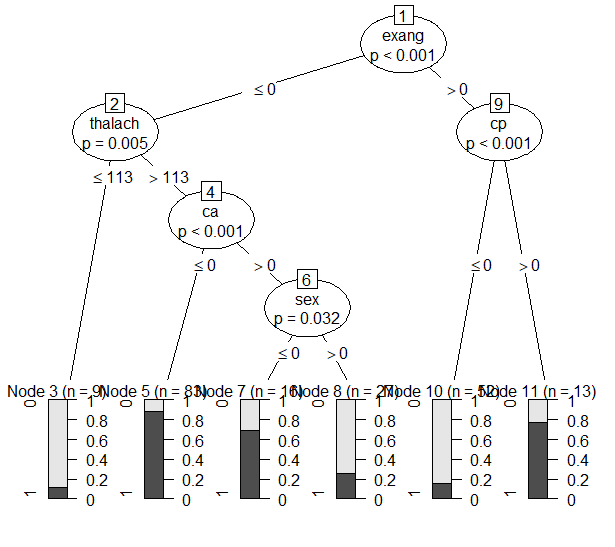
Figure 14 mtry = 5
| 구분 | TPR | Precision | TNR | Accuracy | BCR | F1-Measure |
|---|---|---|---|---|---|---|
| mtry = 3 | 0.690909 | 0.826087 | 0.833333 | 0.757282 | 0.758787 | 0.752475 |
| Mtry = 4 | 0.836364 | 0.766667 | 0.708333 | 0.776699 | 0.769691 | 0.8 |
| mtry = 5 | 0.872727 | 0.761905 | 0.6875 | 0.786408 | 0.774597 | 0.813559 |
- Mtry를 3으로 설정했을 때에는 3번의 split과 4개의 terminal node가 있었다. Minsplit을 50으로 설정했을 때와 결과가 비슷했지만 node 6의 impurity가 거의 0에 가까워졌다. 성능은 accuracy, BCR, F1 모두 0.75 근처였지만 TPR이 0.7 이하라는 비교적 낮은 값을 나타냈다.
- Mtry를 4로 설정했을 때는 8번의 split을 통해 9개의 leaf node가 생성되었다. Leaf node의 개수가 늘어나면서 accuracy, BCR, F1은 개선되었지만 Precision과 TNR이 줄어들었다.
- Mtry를 5로 설정했을 때는 5번의 split을 통해 6개의 leaf node가 생성되었다. 각 node의 impurity도 준수했다. 성능 지표를 살펴보면 precision은 mtry=4인 모델모다 살짝 낮았지만 크게 차이나지 않았고, TNR을 제외한 나머지 지표들에서 성능 향상이 있었다.
3.2.3 Best Model 선정
| 구분 | TPR | Precision | TNR | Accuracy | BCR | F1-Measure |
|---|---|---|---|---|---|---|
| minsplit = 10 | 0.763636 | 0.763636 | 0.729167 | 0.747573 | 0.746203 | 0.763636 |
| minsplit = 50 | 0.690909 | 0.745098 | 0.729167 | 0.708738 | 0.70978 | 0.716981 |
| minsplit = 100 | 0.454545 | 0.757576 | 0.833333 | 0.631068 | 0.615457 | 0.568182 |
| mtry = 3 | 0.690909 | 0.826087 | 0.833333 | 0.757282 | 0.758787 | 0.752475 |
| Mtry = 4 | 0.836364 | 0.766667 | 0.708333 | 0.776699 | 0.769691 | 0.8 |
| mtry = 5 | 0.872727 | 0.761905 | 0.6875 | 0.786408 | 0.774597 | 0.813559 |
모델들을 비교해보았을 때, mtry = 5인 모델이 가장 우수하다고 판단했다. 우선 Accuracy, BCR, F1이 모두 가장 높았다. TPR 측면에서도 가장 좋은 성능을 나타냈다. Precision은 1위는 아니었으나 1위인 0.766667과 크게 차이나지 않아 best model로 선정하기에 무리가 없다고 판단했다. TNR은 가장 낮은 0.6875였으나 위에서 서술했듯이 환자가 아닌 사람을 환자라고 예측하는 것보다 환자를 제대로 예측하는 것이 중요하기 때문에 F1 measure가 높은 mtry = 5 모델을 best model로 선정하였다.
4. “evtree”Package
4.1“evtree”패키지의 옵션
“evtree” 패키지의 ctree 함수가 Classification Tree를 학습할 때 사용자가 지정할 수 있는 옵션의 종류와 의미는 다음과 같습니다.
evtree 함수: evtree(formula, data, subset, na.action, weights, control = evtree.control(…), …)
옵션|의미
—|—
controls| Evtree.control을 통해 tree 구축의 세부적 사항을 설정한다.
minsplit: split하기 전 필요한 weight 합의 최소값
minbucket: leaf node의 weight 합의 최소값
maxdepth: 최종 tree의 최대 depth
ntrees: population의 tree 개수
alpha: cost function의 complexity part를 조절한다. Alpha가 커지면 tree size가 줄어든다.
4.2 옵션의 변화에 따른 Classification Tree들의 차이점 및 Best Model 선정
4.2.1 alpha = 0.5, 1, 2
Alpha를 0.5, 1, 2로 변화시켜가며 tree를 그려보았다.
#4. Evtree package________________________________________________________-
library(evtree)
#4.2.1 alpha = 0.5, 1, 2
Perf.Table <- matrix(0, nrow = 1, ncol = 6)
colnames(Perf.Table) <- c("TPR", "Precision", "TNR", "Accuracy", "BCR", "F1-Measure")
for (i in c(0.5, 1, 2)){
heart.evtree <- evtree(heart.target ~.,data = heart.trn,
control = evtree.control(alpha = i))
plot(heart.evtree)
# Prediction
heart.prey <- predict(heart.evtree, heart.tst, type = "response")
heart.cfm <- table(heart.tst$heart.target, heart.prey)
heart.cfm
Perf.Table<- rbind(Perf.Table, perf_eval(heart.cfm))
Perf.Table
}
Perf.Table
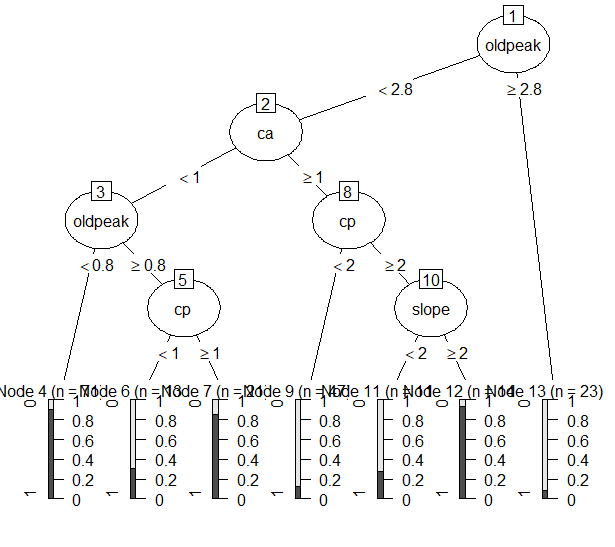
Figure 15 alpha = 0.5
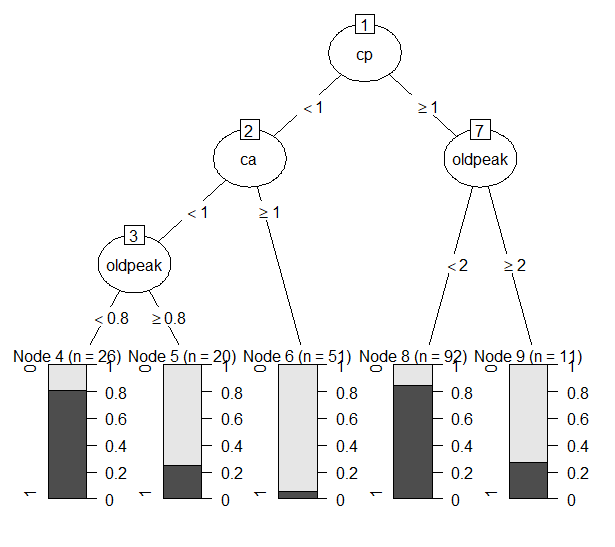
Figure 16 alpha = 1
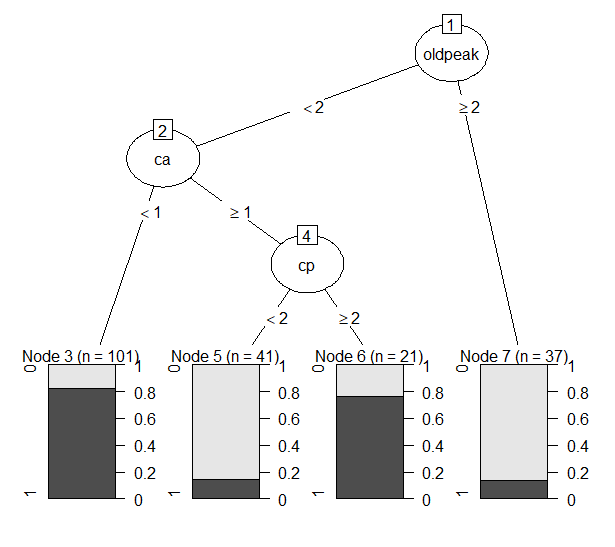
Figure 17 alpha = 2
| 구분 | TPR | Precision | TNR | Accuracy | BCR | F1-Measure |
|---|---|---|---|---|---|---|
| alpha = 0.5 | 0.890909 | 0.830508 | 0.791667 | 0.84466 | 0.839823 | 0.859649 |
| alpha = 1 | 0.818182 | 0.775862 | 0.729167 | 0.776699 | 0.772393 | 0.79646 |
| alpha = 2 | 0.836364 | 0.741935 | 0.666667 | 0.757282 | 0.74671 | 0.786325 |
- Alpha가 커질수록 tree들은 단순해지는 경향을 보였다.
- Alpha = 0.5일 때, leaf node 개수는 가장 많은 7개였다. Node 11을 제외하면 나머지 노드들의 impurity도 비교적 낮은 편이었다. 사용된 변수들은 oldpeak, ca, cp, slope, thal이었다. 모든 performance measure에서 다른 모델보다 성능이 좋다는 것을 확인할 수 있었다.
- Alpha = 1일 때, leaf node 개수는 5개로 줄어들었다. Node들의 impurity는 낮은 편이었다. Split에 사용된 변수들은 cp, ca, oldpeak였다. 위에서는 oldpeak이 가장 먼저 사용된 반면, 여기서는 cp가 가장 먼저 사용되었다. Performance measure를 살펴보면, alpha = 0.5일 때보다 성능이 떨어짐을 확인할 수 있었다.
- Alpha = 2일 때, leaf node 개수는 다시 줄어들어 4개가 되었다. 사용된 변수들은 oldpeak, ca, cp였다. 모든 tree에서 이 세가지 변수가 반복적으로 사용된 것을 보아 분류에 있어 매우 중요한 변수들이라고 판단된다. Performance measure는 alpha = 1일 때보다도 감소해 분류 성능이 떨어졌다는 것을 알 수 있다. 제대로 예측하기에 leaf node의 개수가 너무 적은 것으로 보인다.
4.2.2 ntrees = 11, 15, 20
다음으로 ntrees를 11, 15, 20으로 바꾸어가며 tree를 그려보았다. Ntrees는 최소값이 11이었다.
#4.2.2 ntrees = 11, 15, 20
Perf.Table <- matrix(0, nrow = 1, ncol = 6)
colnames(Perf.Table) <- c("TPR", "Precision", "TNR", "Accuracy", "BCR", "F1-Measure")
for (i in c(11,15,20)){
heart.evtree <- evtree(heart.target ~.,data = heart.trn,
control = evtree.control(ntrees = i))
plot(heart.evtree)
# Prediction
heart.prey <- predict(heart.evtree, heart.tst, type = "response")
heart.cfm <- table(heart.tst$heart.target, heart.prey)
heart.cfm
Perf.Table<- rbind(Perf.Table, perf_eval(heart.cfm))
Perf.Table
}
Perf.Table
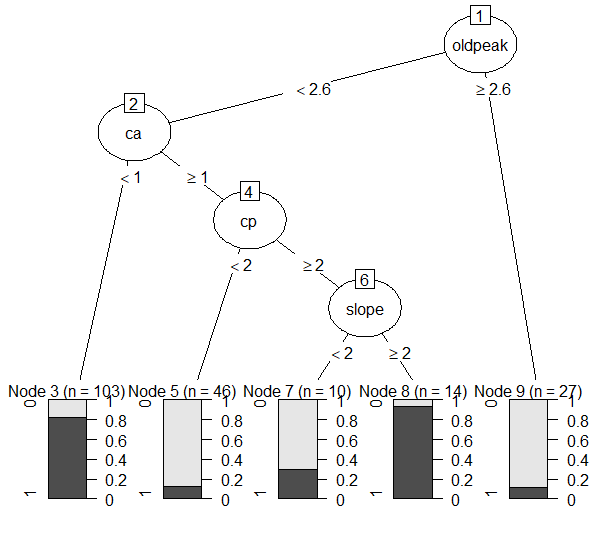
Figure 18 ntrees = 11
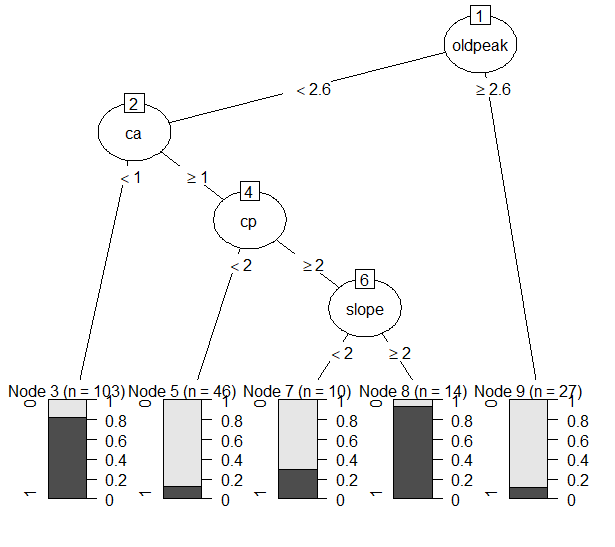
Figure 19 ntrees = 15
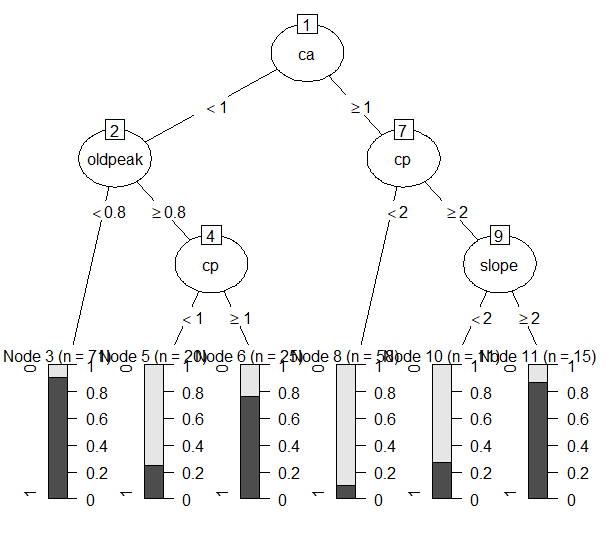
Figure 20 ntrees = 20
| 구분 | TPR | Precision | TNR | Accuracy | BCR | F1-Measure |
|---|---|---|---|---|---|---|
| ntrees = 11 | 0.818182 | 0.775862 | 0.729167 | 0.776699 | 0.772393 | 0.79646 |
| ntrees = 15 | 0.836364 | 0.741935 | 0.666667 | 0.757282 | 0.74671 | 0.786325 |
| ntrees = 20 | 0.818182 | 0.775862 | 0.729167 | 0.776699 | 0.772393 | 0.79646 |
- Ntrees의 최소값이 11이었기 때문이 11부터 증가시켜가며 변화를 살펴보았다.
- Ntrees를 11로 설정했을 때에는 5개의 leaf node가 생성되었고, 각 노드의 impurity는 낮은 편이었다. 사용된 변수들은 oldpeak, ca, cp였다. Ntrees를 15로 설정했을 때는 4개의 leaf node가 생성되었다. 사용된 변수는 마찬가지로 oldpeak, ca, cap였다. 마지막으로 ntrees를 20으로 설정했을 때는 leaf node가 5개 생성되었고, ntrees를 10으로 설정했을 때와 같은 tree가 생성되었다.
- Performance measure를 비교해보면 ntrees = 11과 20에서는 TPR을 제외하면 나머지 measure들에서 모두 ntrees = 15보다 좋은 성능을 나타냈다. Ntrees = 15는 TPR에서만 더 뛰어난 성능을 보였는데, TNR이 0.6667로 낮은 수치를 나타냈고, F1이 다른 모델들보다 떨어지므로 좋은 모델이 아니라고 판단했다.
4.2.3 Best Model 선정
| 구분 | TPR | Precision | TNR | Accuracy | BCR | F1-Measure |
|---|---|---|---|---|---|---|
| alpha = 0.5 | 0.890909 | 0.830508 | 0.791667 | 0.84466 | 0.839823 | 0.859649 |
| alpha = 1 | 0.818182 | 0.775862 | 0.729167 | 0.776699 | 0.772393 | 0.79646 |
| alpha = 2 | 0.836364 | 0.741935 | 0.666667 | 0.757282 | 0.74671 | 0.786325 |
| ntrees = 11 | 0.818182 | 0.775862 | 0.729167 | 0.776699 | 0.772393 | 0.79646 |
| ntrees = 15 | 0.836364 | 0.741935 | 0.666667 | 0.757282 | 0.74671 | 0.786325 |
| ntrees = 20 | 0.818182 | 0.775862 | 0.729167 | 0.776699 | 0.772393 | 0.79646 |
Best model은 alpha = 0.5로 설정한 모델이었다. 모든 성능 지표에서 다른 모델들보다 월등함을 나타냈다. 특히 TPR은 0.89라는 매우 높은 값을 나타냈으며 F1 역시 0.8696으로 다른 모델들에 비해 훨씬 높았다.
5. 각 패키지의 classification tree 비교
5.1 각 패키지에서 제공하는 Tree plot 비교
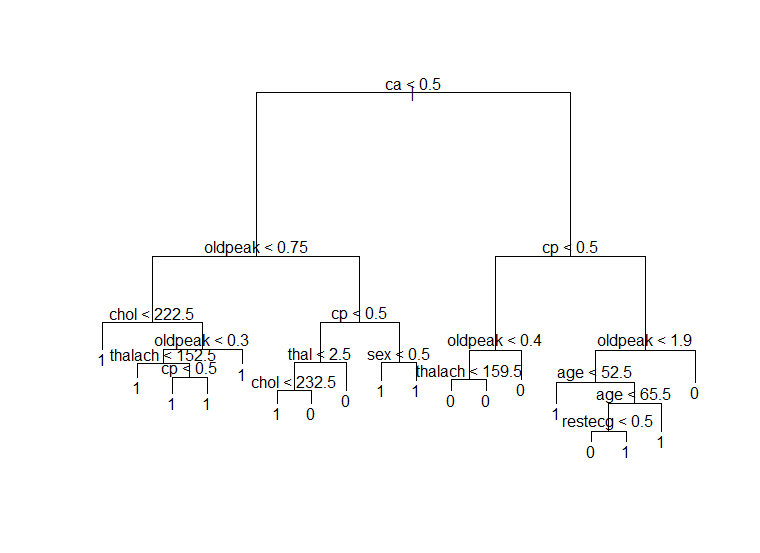
- 위의 tree plot들은 각 패키지에서 제공하는 tree plot이다. tree패키지의 경우 가장 단순한 plot을 그려준다. Decision node에서 사용된 변수는 무엇인지, decision point는 무엇인지 알려주지만 yes or no가 어느 방향인지 알려주지 않기 때문에 해석에 어려움이 있다. 또한 leaf node에서 제공하는 정보가 클래스 정보 밖에 없다.
-
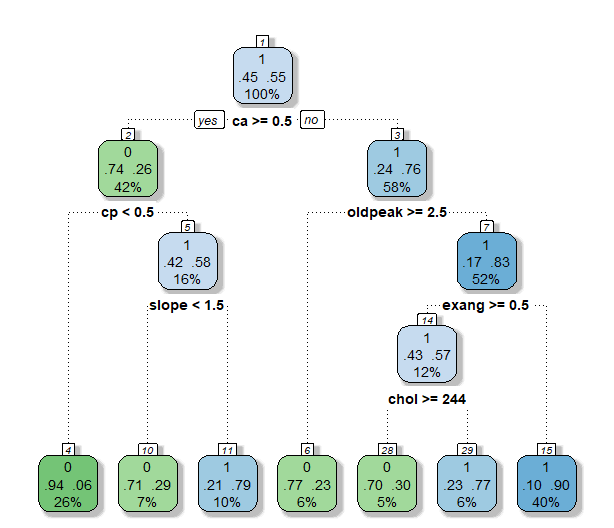
Rpart 패키지로 그린 plot은 보다 많은 정보를 제공한다. 우선 decision node에서 사용된 변수가 무엇인지 뿐만 아니라 각 노드에 전체의 몇 퍼센트에 해당하는 데이터가 들어있는지 알려준다. 더 나아가 각 노드에서 각 클래스의 비중이 어떻게 되는지 알 수 있다. 예를 들어 4번 leaf node를 보면 94%의 데이터가 0이고, 6%의 데이터가 1이므로 이 노드는 0이 할당되었음을 알 수 있다. 또한 여기에는 전체의 26%에 해당하는 데이터가 포함된다. Leaf node에서 impurity는 색깔로 나타내어 주는데, 0에 가까울수록 초록색이고 1에 가까울수록 파란색이다. 이를 통해 진한 파란색과 녹색은 impurity가 낮고, 하늘색은 impurity가 높음을 알 수 있다.

-
Evtree 패키지로 그린 tree plot은 decision node에서 어떤 decision variable이 사용되었는지 알려준다. Decision point는 arc 위에 쓰여 있다. rpart에서는 impurity 정도를 색깔로 나타내었다면 evtree 패키지에서는 바그래프로 나타낸다. 검정색의 비중이 높을수록 1에 가까움을 나타낸다. Rpart와 tree 패키지에서는 각 leaf node가 어떤 클래스로 할당되었는지도 나타내지만 evtree 패키지는 알려주지 않는다는 단점이 있다. Rpart는 각 노드에 할당된 데이터가 전체의 몇 퍼센트인지 비율로써 나타내지만 evtree 패키지에서는 n이 얼마인지 수치로 나타내어 준다는 차이점이 있다.
5.2 각 패키지에서 제공한 Classification tree들의 분류 성능 비교 및 논의
| 구분 | TPR | Precision | TNR | Accuracy | BCR | F1-Measure |
|---|---|---|---|---|---|---|
| Tree | 0.8181818 | 0.7894737 | 0.75 | 0.7864078 | 0.7833495 | 0.8035714 |
| Rpart | 0.8727273 | 0.8 | 0.75 | 0.815534 | 0.8090398 | 0.8347826 |
| Party | 0.872727 | 0.761905 | 0.6875 | 0.786408 | 0.774597 | 0.813559 |
| Evtree | 0.890909 | 0.830508 | 0.791667 | 0.84466 | 0.839823 | 0.859649 |
각 패키지에서 선정한 best model에 대한 performance measure를 비교해보았다.
Tree 패키지로 구축한 모델은 pruning을 수행한 후의 모델로 선정했다. 이 모델은 대부분의 performance measure 측면에서 가장 낮은 수치를 나타냈다. BCR과 TNR의 경우 Party보다는 나은 성능을 보였지만 다른 지표들에서 좋지 않은 성능을 나타냈다. Rpart로 구축한 모델은 비교적 우수한 성능을 나타냈다. Party로 구축한 모델은 TPR이 Rpart와 비슷했지만 나머지 지표들에서 훨씬 성능이 떨어지는 것을 보였다. 특히 TNR의 경우 0.6875로 다른 모델들에 비해 현저히 낮은 수치를 나타냈다. 하지만 Accuracy는 tree패키지와 비슷하게 나타났는데, 이는 TPR이 tree에 비해 훨씬 높았기 때문으로 보인다. 마지막으로 evtree 패키지로 구축한 tree는 모든 performance measure에서 가장 좋은 성능을 나타냈다. 특히 TPR의 경우 0.9에 가까운 수치로 다른 모델들에 비해 월등했다. Precision, Accuracy, BCR, F1 measure 모두 0.8 이상으로 높은 편이었다.
이렇게 모델들을 비교해 보았을 때, Evtree로 구축된 다음 모델이 best model이었다.
Figure 21 Evtree Best model
